Opciones terapéuticas actuales IIb/V: Terapia Antisentido (06/2012)
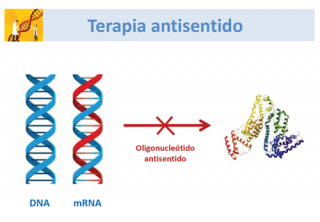
La terapia antisentido se basa en el uso de oligonucleótidos (pequeños fragmentos de ácido nucleico (13-25 nucleótidos), antisentido, complementarios para una secuencia específica de un gen, con objeto de unirse al mRNA del mismo y evitar así la producción de una proteína mutada.
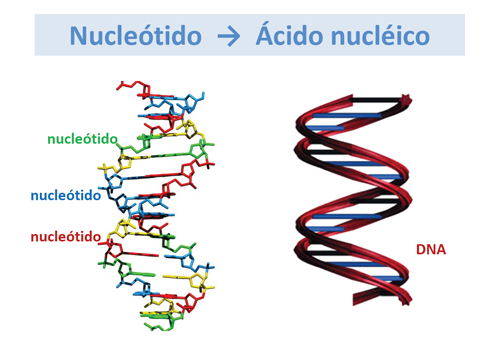
¿Qué son los nucleótidos?
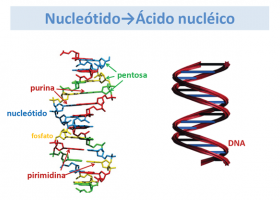
Los nucleótidos son cada una de las unidades que forman el DNA o el RNA. Están formados por una base nitrogenada, una pentosa (azúcar cíclico de 5 átomos de carbono) y un grupo fosfato, unidos por enlaces covalentes (fuertes). Las bases nitrogenadas son purinas (adenina y guanina) y pirimidinas (timina, citosina y uracilo).
Así, existen 5 nucleótidos diferentes en el DNA y RNA: adenina, guanina, timina, citosina y uracilo.
Una larga cadena de muchos nucleótidos constituye un ácido nucleico. En el ácido desoxiribonucléico (DNA) la pentosa es la desoxiribosa, mientras que en el ácido ribonucléico (RNA) es la ribosa. Ambos ácidos (DNA y RNA) contienen la información genética que codifica para la síntesis de todas las proteínas celulares.
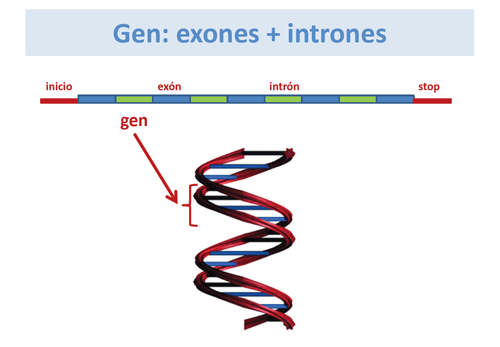
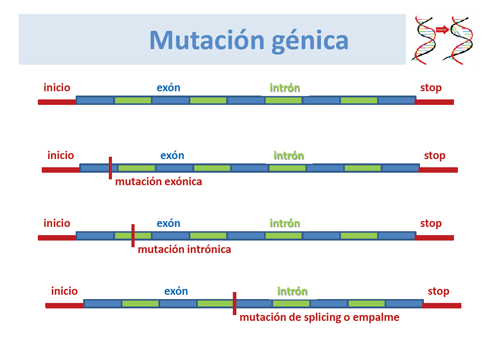
Un gen está formado por una larga cadena de DNA, en la que se distinguen exones e intrones. Los exones son las regiones codificantes que van a proporcionar la información para la síntesis de una proteína, mientras que los intrones son regiones no codificantes, que se hallan intercaladas en el gen.
¿Cómo se produce la transferencia de la información contenida en el DNA para la formación de proteínas?
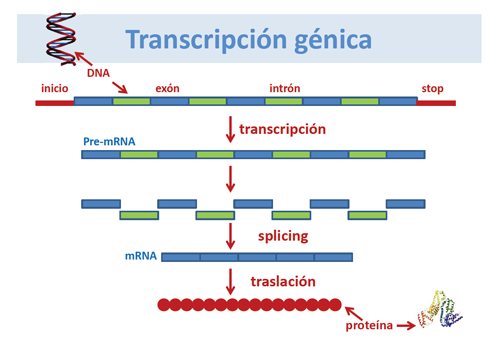
En forma muy resumida, ya que este proceso es muchísimo más complejo, esta transferencia de información desde el DNA hasta la formación de una proteína o proceso de transcripción génica se realiza mediante diversas formas de RNA.
Las secuencias de los exones del DNA se transcriben para formar el RNA mensajero (mRNA). A una determinada base púrica o pirimidínica del DNA le corresponde otra complementaria del RNA), de forma que se van disponiendo una a una formando una cadena a medida que “lee” el DNA.
De esta forma el RNA contiene la información génica del DNA necesaria para la formación de la proteína. Mediante una maquinaria celular posterior, la cadena de RNA da lugar a la formación de una cadena de aminoácidos, es decir, a una proteína.
En el proceso de formación del mRNA se eliminan los intrones del DNA, en un proceso de empalmes o splicing, manteniéndose en el mRNA sólo la información de los exones del DNA, que codifican para la proteína.
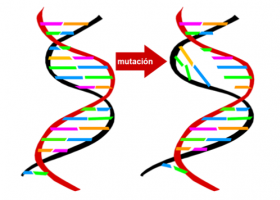
¿Qué pasa cuando hay un cambio o mutación en la secuencia del DNA?
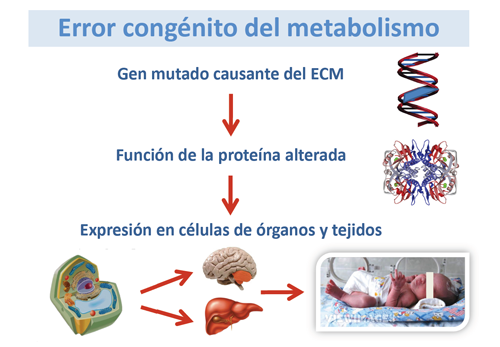
Una mutación en un gen dará lugar a una proteína anómala. Si su función era importante en el metabolismo dará lugar a un error congénito del metabolismo (ECM).
¿Las mutaciones se producen en los exones o también en los intrones del DNA?
Las mutaciones se pueden producir en cualquier punto de la secuencia del DNA, con diferentes consecuencias según su mayor o menor efecto sobre la síntesis de la proteína.
Cuando ocurre una inserción (se incluye un nucleótido que no debería estar en la cadena normal de DNA) o deleción (falta algún/os nucleótidos que deberían estar en la cadena normal de DNA) de uno o más nucleótidos en una zona de empalme (splicing) de un intrón, puede ocurrir que uno o más intrones pueden no ser eliminados y entonces se expresan en el mRNA como si se tratase de exones, dando lugar a proteínas anómalas por mutaciones de splicing.
¿Son frecuentes las mutaciones de splicing causantes de ECM?
Constituyen alrededor de un 10% de las mutaciones descritas en las bases de datos de genes humanos. Algunas se hallan en los puntos de unión exón-intrón y otras en el interior de los intrones, lo que puede dar lugar a la aparición de secuencias anómalas de intrones en el mRNA o pseudoexones (falsos exones), con graves consecuencias en la formación de la proteína.
Se han descrito con frecuencia en acidemias orgánicas, enfermedades lisosomales, defectos congénitos de la glicosilación y deficiencias de tetrahidrobiopterina. Tienen el inconveniente de que, en ocasiones, no se detectan en un análisis mutacional de rutina, por lo que pueden pasar desapercibidas y complicar el estudio genético de los pacientes que las portan.
Recientemente se está estudiando el uso de oligonucleótidos antisentido para corregir la función génica eliminando la secuencia intrónica o pseudoexón.
¿Qué son los oligonucleótidos?
Son pequeños fragmentos de ácido nucleico (13-25 nucleótidos), que pueden usarse como agentes terapéuticos o bien para estudiar la función de un gen.
¿Por qué se llaman oligonucleótidos antisentido?
Porque están diseñados para que sean complementarios a una secuencia específica de un gen, es decir, para que sean capaces de unirse a una determinada secuencia génica del RNA mensajero (mRNA), evitando con ello la transcripción de la información anómala, es decir, que se produzca una proteína anómala.
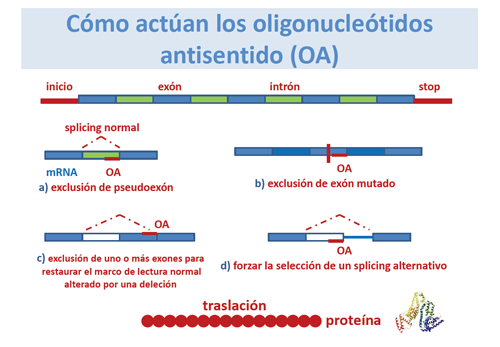
¿Cómo actúan los oligonucleótidos antisentido?
Pueden actuar:
a) Excluyendo un pseudoexón debido a una mutación en el punto de empalme exón-intrón.
b) Induciendo la “eliminación” o “silenciamiento” de exones que contienen una mutación.
c) Saltándose uno o más exones que flanquean una deleción de forma que se altera la lectura sorteando la zona mutada.
d) Forzando la selección de un punto de splicing alternativo.
¿Qué problemas presenta la Terapia Antisentido?
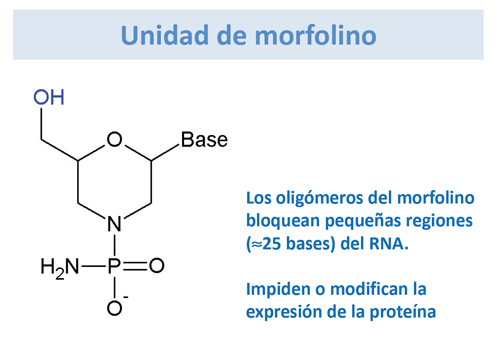
- Los oligonucleótidos presentan inestabilidad debida a la rápida degradación por nucleasas (enzimas que existen en células normales cuya función es digerir cadenas de ácidos nucleicos). Este problema se ha ido superando con la aparición de oligonucleótidos modificados, como los derivados del morfolino, que son muy estables “in vivo”, muestran gran afinidad hacia las dianas y potente actividad biológica.
- La liberación de los oligonucleótidos antisentido requiere sistemas de transporte que sean capaces de liberarlos intracelularmente, donde deben hacer su función. Algunos de ellos, como los péptidos, pueden provocar una respuesta inmune (activación del sistema inmune que nos defiende de agentes potencialmente nocivos) que debe evitarse. El tratamiento se realiza mediante una administración continuada, así que esta reacción inmune debe ser evitada.
- La mayoría de los oligonucleótidos antisentido acaban acumulándose en el hígado y en el riñón, lo que es importante para los ECM que se expresan principalmente en estos órganos, como las acidurias orgánicas, aminoacidopatías, enfermedades lisosomales y peroxisomales.
- Hay que considerar el posible efecto tóxico de liberar oligonucleótidos en dosis altas y repetidas a largo plazo, ya que los pacientes se deben tratar a lo largo de sus vidas.
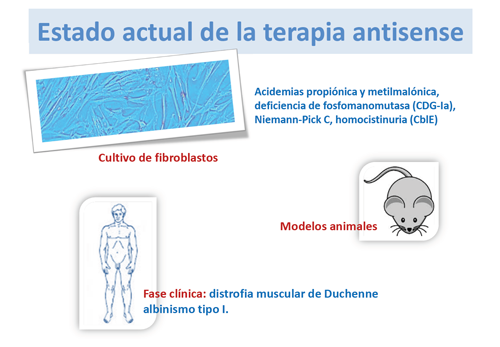
¿Cuál es el estado actual de dicha terapia?
Se ha aplicado con éxito en líneas celulares de pacientes en cultivo y en modelos animales en diferentes enfermedades metabólicas.
En la distrofia muscular de Duchenne se está aplicando en fase clínica en el humano, aunque su administración se realiza directamente en el músculo.
Se requieren oligonucleótidos de nueva generación con mejoras en la afinidad por la diana, mayor bioestabilidad y farmacocinética (vida media en el cuerpo humano, velocidad de eliminación…) optimizada. También se están investigando en enfermedades infecciosas y cáncer.
¿Qué ventajas e inconvenientes tiene la Terapia Antisentido?
El mRNA se transcribe en su contexto natural y bajo los mecanismos celulares de control de la expresión normales. En este aspecto, la intervención es más fácil que en el caso de la terapia génica clásica.
Por otra parte, el hecho de que se base en la modulación del splicing hace que la terapia antisentido sea específica de un gen y de un tipo de mutación, lo que requiere un desarrollo y optimización individualizados.
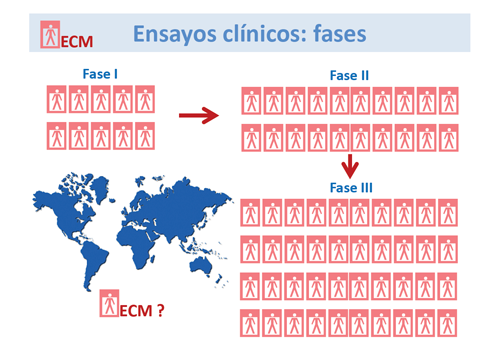
Considerando que los ECM son enfermedades minoritarias, esto dificulta las fases de ensayos clínicos en los que se necesita un gran número de individuos. Se requieren estudios multicéntricos para la fase III de ensayo clínico.
Dras. Mercedes Serrano y Mª. Antonia Vilaseca (Equipo Guía metabólica), con la supervisión especial de la Dra. Belén Pérez-González, experta en terapia antisentido (Centro de Diagnóstico de Enfermedades Moleculares, Centro de Biología Molecular, CIBERER, Universidad Autónoma de Madrid, España). Resumen basado en el artículo: Pérez B et al. J Inherit Metab Dis (2010) 33:397–403.
- 17910 lecturas