Nuevas tecnologías en el diagnóstico y seguimiento de las enfermedades metabólicas hereditarias

El análisis de aminoácidos en diferentes líquidos biológicos es esencial para el diagnóstico de diversos errores congénitos del metabolismo (ECM). Además, los aminoácidos pueden servir como sensibles biomarcadores para el estudio de otras enfermedades.
Hoy en día se ha caracterizado genéticamente un amplio número de ECM relacionados con alteraciones del metabolismo de los aminoácidos, siendo algunos de ellos enfermedades tratables.
Por otra parte, aun cuando algunas aminoacidopatías se pueden detectar mediante los programas de cribado neonatal, no todos los aminoácidos son detectables en ellos, por lo que el desarrollo y adaptación de nuevos procedimientos bioquímicos, rápidos y sensibles, para el diagnóstico y seguimiento de ECM en diferentes muestras biológicas es sumamente importante.
Tradicionalmente la determinación de aminoácidos se había realizado por otros procedimientos (cromatografía de intercambio iónico, cromatografía líquida de alta resolución) que, aunque permitían la separación de la mayor parte de aminoácidos fisiológicos, tenían algunos inconvenientes, como el largo tiempo de separación de los aminoácidos (2-3 horas por muestra), elevado volumen de muestra necesario para el análisis (200-300 microlitros), detección de otros compuestos que podían interferir en el análisis.
Algunos requerían una derivatización de la muestra que daba lugar a productos derivados inestables.
En este artículo se explica detalladamente la adaptación de un nuevo procedimiento de cromatografía líquida de ultra-alta resolución acoplado a espectrometría de masas (UPLC-MS/MS) para el análisis de aminoácidos en diferentes líquidos biológicos.
La cromatografía líquida permite separar los distintos aminoácidos, mientras que la espectrometría de masas permite su identificación y cuantificación.
El proceso completo de análisis consta de:
- Preparación de las muestras para el análisis (fase preanalítica)
- Derivatización de los compuestos a analizar (aminoácidos y compuestos relacionados)
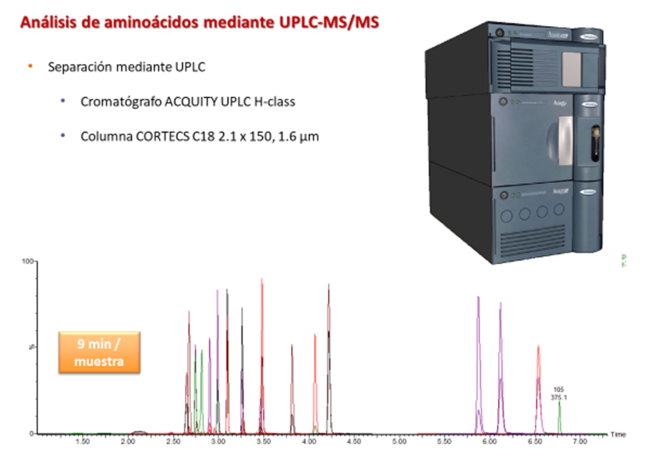
- Separación de sustancias mediante UPLC
Preparación de las muestras biológicas
Las muestras biológicas se deben tratar previamente a su análisis. Debe añadirse un estándar interno (SI), que es una sustancia química que se añade a la muestra en una concentración constante y permite la cuantificación de los compuestos que se van a analizar, corrigiendo las posibles pérdidas.
En el caso de plasma y orina deben eliminarse las proteínas (desproteinizar) mediante un derivado del ácido fórmico. El LCR se puede analizar directamente.
Los aminoácidos en sangre seca deben extraerse previamente durante una hora mediante ácido tricloroacético (ver pauta en la figura adjunta).

Derivatización de los aminoácidos
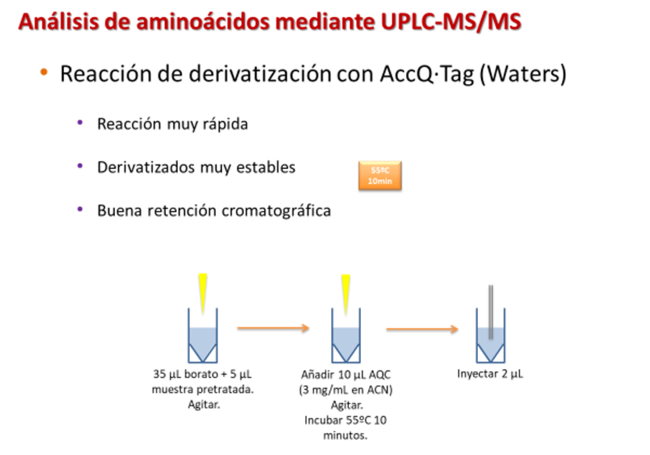
La muestra debe tratarse con AccQ·Tag (Waters) para derivatizar los aminoácidos a analizar.
Separación y detección mediante UPLCMS/MS

Resultados
Validación analítica
Todos los aminoácidos se han separado en menos de 7 minutos, siendo el tiempo total de análisis por muestra de 9 minutos.
Se han optimizado las condiciones analíticas para poder separar los isómeros (compuestos formados por los mismos elementos en iguales proporciones, pero con diferente estructura molecular) de algunos aminoácidos (por ejemplo: leucina, isoleucina y aloisoleucina, o bien, sarcosina, β-alanina y alanina, o bien ácidos α-, β- y γ-aminobutírico).
Muestras de pacientes analizadas
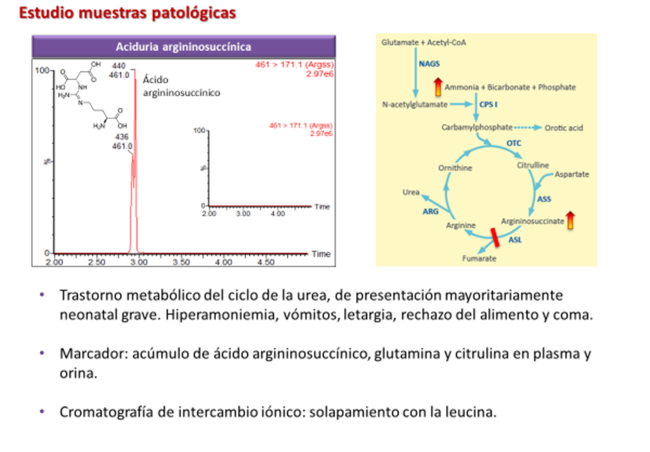
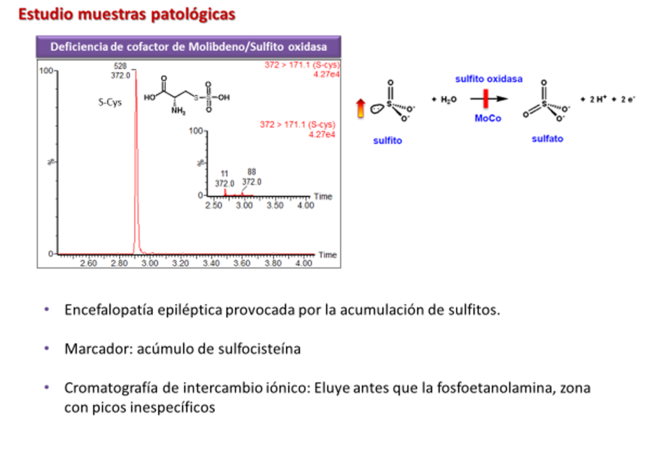
Se han analizado 90 muestras de plasma, orina y líquido cefalorraquídeo (LCR) de pacientes con diferentes ECM (aminoacidopatías y otras enfermedades relacionadas): enfermedad de jarabe de arce, fenilcetonuria, hiperlisinemia, aspartilglucosaminuria, deficiencia de antiquitina, síndrome de hiperamonemia-hiperornitinemia-homocitrulinemia, aciduria argininsuccínica, deficiencia de ornitina transcarbamilasa, citrulinemia tipo 1, argininemia, lisinuria con intolerancia a proteínas, deficiencia de prolidasa, deficiencia de cofactor molibdeno, tirosinemia, deficiencia de la quinasa de la deshidrogenasa de los cetoácidos ramificados, hiperglicinemia no-cetósica y homocistinuria clásica.
También se han analizado muestras de sangre seca de fenilcetonuria, enfermedad de jarabe de arce, tirosinemia y muestras control).
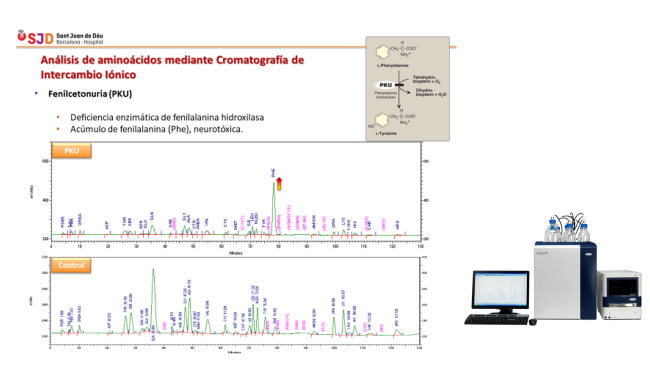
El análisis de estas muestras ha confirmado los resultados alterados de aminoácidos que habían sido ya analizados previamente por cromatografía de intercambio iónico (método tradicional), comprobándose así que hubieran permitido su detección y cuantificación.
Valores de referencia
Para calcular los valores de referencia (valores normales) se han usado 60 muestras de plasma, 80 de orina y 40 de LCR de sujetos control, sin enfermedad aparente.
Se ha comprobado que algunos aminoácidos variaban con la edad, lo que ha permitido establecer diferentes grupos de edad.
Conclusiones
Se ha desarrollado y validado un procedimiento rápido (9 minutos total por muestra) y preciso para la cuantificación de 25 aminoácidos, y la de 17 compuestos relacionados en plasma, orina, LCR y sangre seca.
El tratamiento previo de las muestras es simple, fácil y rápido. El procedimiento resulta reproducible, sensible y fiable. El volumen de muestra requerido es mínimo (25 microlitros). La separación de isómeros es efectiva y rápida.
La comparación de métodos con el tradicional de cromatografía de intercambio iónico es buena, así como también los resultados del control de calidad externo (ERNDIM).
Permite cuantificar algunos compuestos no detectables por el método tradicional: sulfocisteína (marcador de la deficiencia de cofactor molibdeno), ácido pipecólico (marcador de la deficiencia de antiquitina), ácido argininsuccínico (marcador de la acidemia argininsuccínica), aspartilglucosamina (marcador de la aspartilglucosaminuria), glicilprolina (marcador de la deficiencia de prolidasa).
Resumen del articulo: “A targeted metabolomic procedure for amino acid analysis in different biological specimens by ultra-high-performance liquid chromatography–tandem mass spectrometry” publicado on line en: Metabolomics (2018) 14:76 por M. Casado, C. Sierra, M. Batllori, R. Artuch, y A. Ormazabal.
- 1551 lecturas



