El mapa genético de las enfermedades mitocondriales en España

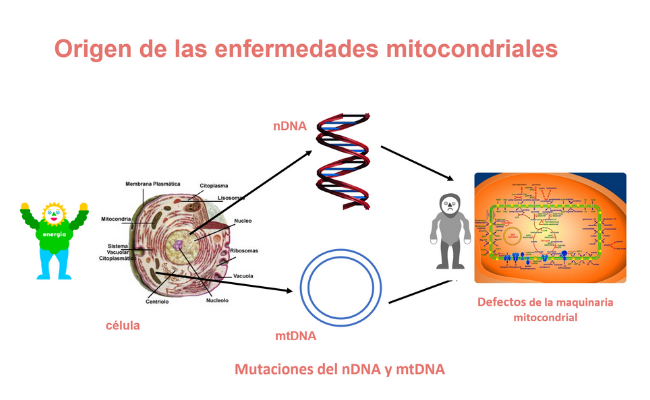
Las enfermedades mitocondriales (EM) están causadas por mutaciones en el ADN nuclear (nDNA) o ADN mitocondrial (mtDNA). Aunque la población pediátrica es, probablemente la más afectada, la edad de presentación varía entre los pacientes y la aparición en adultos es relativamente frecuente.
Todos los tipos de herencia son posibles (esporádica, materna, ligada al cromosoma X y autosómica dominante y recesiva).
El diagnóstico de las EM requiere un enfoque integral que comprenda la exploración clínica de diferentes órganos y sistemas, imagen, anatomía patológica, electrofisiología, exámenes bioquímicos y genética.
Se habían publicado algunos estudios sobre la prevalencia de EM en áreas geográficas específicas de diferentes países. Por ejemplo, en el noroeste de España se describió una prevalencia de 8 por 100.000 en la población pediátrica y 5,7 por 100.000 en la población adulta (Castro-Gago y col, 2006), pero los criterios de inclusión eran más amplios, incluyendo pacientes sin diagnóstico genético definitivo. Recientemente, los criterios diagnósticos para EM se han revisado (Witters y col, 2018).
Por ello, en este estudio solo se han considerado pacientes con un diagnóstico molecular definitivo basado en la presencia de mutaciones patógenas en el nDNA o mtDNA, que afectan a diferentes funciones mitocondriales.
Objetivo del estudio
Como primer paso para el desarrollo de un registro de pacientes con EM en España, se recogió todos los casos diagnosticados durante el período de 1990 a 2020 en hospitales y centros de referencia a nivel nacional. Solo se incluyeron pacientes que albergaran mutaciones patógenas en el mtDNA o nDNA.
Materiales y métodos
Fuente de datos y protocolo de selección de pacientes
Se contactó con todas las unidades clínicas españolas y laboratorios que trabajan en el diagnóstico de EM, en total, 39 hospitales/laboratorios pertenecientes a 49 de 50 provincias españolas. Para cada caso se consideraron las siguientes variables: gen afectado, modo de herencia, variantes patológicas, año y edad de diagnóstico (edad pediátrica o adulta), nombre del médico que solicitó el análisis, y laboratorio genético donde se realizaron las pruebas diagnósticas.
Selección de genes
Se seleccionaron 293 genes mitocondriales con las siguientes funciones biológicas:
- Síntesis de complejos, factores de ensamblaje y portadores de electrones para la fosforilación oxidativa (OXPHOS).
- Mantenimiento del mtDNA, incluida la homeostasis de nucleósidos.
- Expresión de mtDNA que afecta la síntesis, procesamiento y modificación de ARN mitocondrial (ribosómico, de transferencia y mensajero) y biogénesis de ribosomas mitocondriales.
- Biogénesis de cofactores enzimáticos (agrupaciones Fe-S).
- Homeostasis y control de calidad mitocondrial que afectan la importación de proteínas en las mitocondrias, metabolismo de lípidos, morfología mitocondrial (fusión / fisión, organización de crestas mitocondriales), control de calidad de proteínas anormales y mecanismos de apoptosis/autofagia.
- Metabolismo energético relacionado con OXPHOS, incluidos defectos en el ciclo de Krebs, metabolismo del piruvato y transporte de metabolitos específicos.
Finalmente, se seleccionaron 2.761 casos, que se agruparon de diferentes formas: casos pediátricos frente a adultos, y genes mutados en el mtDNA frente a nADN. Para las mutaciones en el mtDNA, solo se incluyeron los casos índices y no sus familiares sin signos clínicos.
Estimación del número de pacientes
Se calculó la incidencia total (número de casos nuevos por año/población española) y la incidencia por grupo de edad (pacientes pediátricos y adultos) según el intervalo de confianza (IC) del 95 %. Las incidencias se expresaron como casos por millón de habitantes durante el período 2014 - 2020, porque durante este período se implementó el uso de técnicas de secuenciación de nueva generación para el diagnóstico de EM, lo que aumentó considerablemente el número de casos diagnosticados.
Diagnóstico genético
Debido al extenso período de reclutamiento, se utilizaron diferentes técnicas genéticas para el diagnóstico. Pero a partir de 2014, el uso de técnicas de secuenciación de nueva generación se fue implementando en la mayoría de los centros de diagnóstico.
Resultados
De los 2.761 pacientes seleccionados:
- 508 pacientes tenían mutaciones en 112 genes del nDNA: 384 eran pacientes pediátricos (75 %), 121 adultos y en 3 casos no se documentó la edad.
- 1.105 pacientes portaban mutaciones en 28 genes codificados por el mtDNA: 367 eran pacientes pediátricos (33 %), 716 adultos, y en 22 casos no se documentó la edad.
- 1.148 casos mostraron mutaciones en el gen MT-RNR1, que se asocian con ototoxicidad a aminoglucósidos y/o hipoacusia no sindrómica: 464 eran pacientes pediátricos (56 %), 366 adultos, y en 318 casos no se documentó la edad.
En cuanto a las mutaciones del nDNA, el gen más frecuentemente mutado en niños fue PDHA1 (que afecta al complejo piruvato deshidrogenasa). En cuanto a los adultos, los genes más mutados (descritos en más de 20 casos) fueron TYMP y TK2, implicados en el mantenimiento del mtDNA. Esto puede ser debido al uso de metabolitos específicos en suero y orina como biomarcadores de EM y también a la existencia de centros de referencia para estas enfermedades específicas en España. Otros genes nucleares frecuentemente mutados en adultos son POLG, OPA1 y TWNKL, también implicados en el mantenimiento del mtDNA.
Para las mutaciones del mtDNA, aparte de la mutación MT-RNR1 m.1555A> G, las mutaciones más frecuentes fueron la m.3243A>G en niños y deleciones en adultos.
Como el número de casos notificados secundarios a mutaciones de nADN aumentaron considerablemente a partir del 2014, debido a la implementación de las tecnologías de secuenciación de nueva generación, se consideró este período para calcular la incidencia. Entre 2014 y 2020, a excepción de los casos de MT-RNR1, la incidencia fue de 6,34 (IC del 95 %: 5,71 a 6,97) casos por millón de habitantes en la edad pediátrica y 1,36 (IC del 95 %: 1,22 a 1,50) para los adultos.
Conclusiones
Este es el primer estudio que aporta datos epidemiológicos a nivel nacional para las EM en España. La aplicación de tecnologías de alto rendimiento en el diagnóstico de rutina de las EM es indispensable para la identificación de un gran número de genes mitocondriales.
Resumen del artículo
Bellusci, M.; Paredes-Fuentes, A.J.; Ruiz-Pesini, E.; Gómez, B.; MITOSPAIN Working Group; Martín, M.A.; Montoya, J.; Artuch, R. The Genetic Landscape of Mitochondrial Diseases in Spain: A Nationwide Call. Genes 2021, 12, 1590.
- 784 lecturas